在國家自然科學基金項目等資助下,中國科學院生物物理研究所徐濤院士和紀偉教授級**工程師在提高光學顯微鏡分辨率技術領域取得重要進展。相關成果以“Molecular Resolution Imaging by Repetitive Optical Selective Exposure”( 基於重複光學選擇曝光的分子分辨率成像技術)為題,於2019年9月9日在Nature Methods(《自然方法學》)雜誌在線發表。論文鏈接://www.nature.com/articles/s41592-019-0544-2。
該工作提出了一種基於激光幹涉條紋定位成像的新技術,並據此研製出新型單分子幹涉定位顯微鏡(Repetitive Optical Selective Exposure, ROSE),將熒光顯微鏡分辨率提升至3 nm以內的分子尺度,單分子定位精度接近1 nm,可以分辨點距為5 nm的DNA origami(DNA 折紙)結構。為降低單分子發光時的閃爍和漂白對亮度和定位精度產生的不良影響,研發團隊對顯微鏡光路進行了創造性地設計,分別為:基於電光調製器的幹涉條紋快速切換激發光路,基於諧振振鏡掃描的6組共軛成像光路,兩種光路的同步實現了高達8 kHz的分時成像,確保在相機的單次曝光時間裏把每個單分子發光狀態均勻分配給6個幹涉條紋,有效避免了熒光分子發光能力波動對定位精度的幹擾。
研發團隊利用該技術對不同熒光位點間距的DNA origami陣列進行驗證測試,證明幹涉成像分辨率達到了3 nm的分子水平。後續的細胞實驗結果顯示,該技術在免疫標記的微管、CCP(clathrin coated pits,網格蛋白有被小窩)以及較致密的細胞骨架成像時展現出良好性能,該工作使得超高分辨光學顯微鏡家族再添新成員,光學顯微鏡分辨率被進一步突破,將為進一步解析精細亞細胞的組分和生物大分子的納米結構提供有力工具。
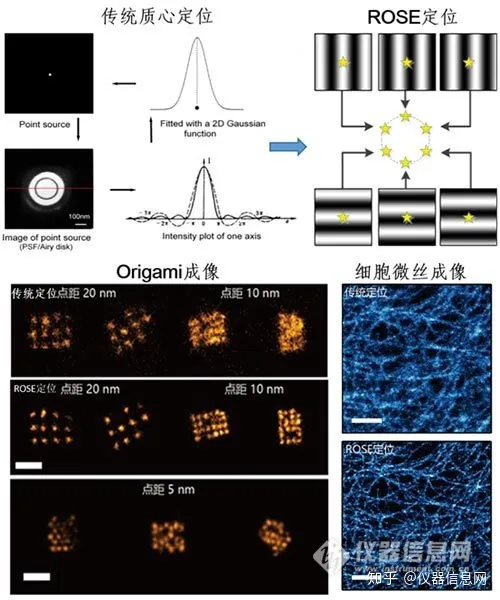
圖. ROSE幹涉定位與傳統質心定位的原理示意,以及用於DNA origami和細胞微絲成像效果比較
[來源:國家自然科學基金委員會]